Untersuchungen zu Identifizierung und Differenzierung von aviären Mykoplasmen mittels Matrix-Assisted Laser Desorption/Ionization Time-of-Flight Massenspektrometrie (MALDI-TOF MS)
Seiten
2020
VVB Laufersweiler Verlag
978-3-8359-6914-8 (ISBN)
VVB Laufersweiler Verlag
978-3-8359-6914-8 (ISBN)
- Keine Verlagsinformationen verfügbar
- Artikel merken
Mykoplasmen sind zellwandlose Bakterien der Klasse Mollicutes, die sowohl beim Nutzgeflügel, als auch bei anderen Vogelspezies beschrieben wurden (Lierz et al. 2008a; Möller Palau-Ribes et al. 2016). Je nach Vogelart und Mykoplasmenspezies spielen sie eine Rolle als Krankheitserreger oder sind Teil der Mikrobiota des Respirationstrakts (Lierz et al. 2008a; Möller Palau-Ribes et al. 2016). Auf Grund der variierenden klinischen Bedeutung sowie des unterschiedlichen Vorkommens verschiedener Mykoplasmenspezies bei Vogelarten, ist die Differenzierung der nachgewiesenen aviären Mykoplasmen von großer Wichtigkeit. Die Identifizierung mit Hilfe konventioneller immunologischer, phänotypischer und molekularbiologischer Methoden ist anspruchsvoll und zeitaufwändig. Die häufig durchgeführten Polymerase-Kettenreaktionen (PCRs) sind meist auf eine bestimmte Spezies zielgerichtet. Mykoplasmen-genusspezifische PCRs erfordern anschließend eine aufwändige Sequenzierung, bei der Untersuchungen aus Probenmaterial zu Fehlidentifikationen führen können. Zudem stehen manche Diagnostika, beispielsweise auf Grund von fehlenden kommerziellen Antiseren für immunologische Methoden oder fehlenden speziesspezifischen PCRs, für viele Mykoplasmenspezies nicht zur Verfügung. Matrix-Assisted Laser Desorption/Ionization Time-of-Flight Massenspektrometrie (MALDI-TOF MS), eine schnelle, verlässliche und kostengünstige Methode zur Differenzierung von Bakterien und Pilzen mit dem Potenzial konventionelle phänotypische Methoden zu ersetzen (Cherkaoui et al. 2010), spielt in der Diagnostik aviärer Mykoplasmen bislang keine Rolle.
In der vorliegenden Arbeit wurde die Anwendbarkeit von MALDI-TOF MS zur Identifizierung und Differenzierung aviärer Mykoplasmen evaluiert. Dazu wurde eigens eine massenspektrometrische Referenzdatenbank erstellt, welche anfänglich 27 MSP aus 23 aviären Mykoplasmenspezies beinhaltete. Nach Erstellung der Datenbank wurden 112 Feldisolate, sowie vier Lebendimpfstoff-Isolate und ein Referenzstamm aviärer Mykoplasmen, deren Spezies bereits mittels genusspezifischer PCR und Sequenzierung des Amplifikats bzw. einer speziesspezifischen PCR und Restriktionsenzymanalyse identifiziert worden waren, mit MALDI-TOF MS und der Mykoplasmen-Referenzdatenbank mit maximal 2 Wiederholungen untersucht.
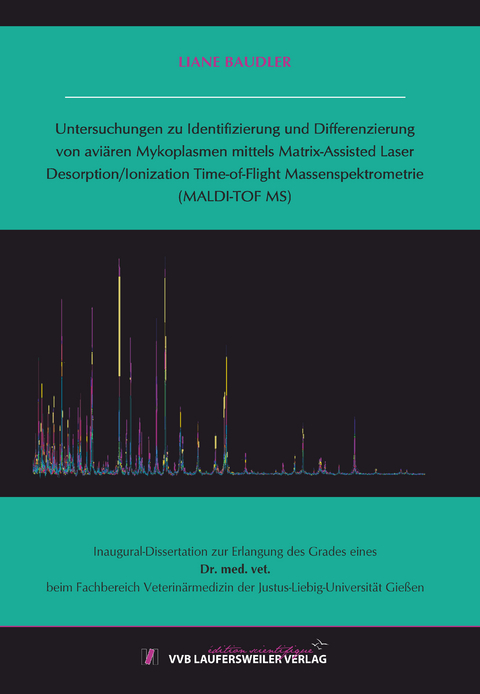
Die MALDI-TOF MS-Analyse wies für 70 % (82/117) der Stämme bzw. Isolate einen Score von ≥ 2,000, für 20 % (23/117) einen Score von ≥1,700 und für 10 % (12/117) Identifizierungsergebnisse mit einem Score von < 1,700 auf. Es zeigte sich, dass besonders die Analysen der Isolate von M. gallisepticum, M. glycophilum, M. gypis, M. synoviae sowie M. sturni zu niedrigen Scores führten. Daraufhin wurde die MSP-Referenzdatenbank auf 36 MSP erweitert und hier besonders die genannten Spezies berücksichtigt. Der erneute Vergleich der bereits gemessenen Spektra mit der erweiterten Datenbank führte zu einer deutlichen Verbesserung der Ergebnisse. Für 96% (112/117) der Isolate bzw. Stämme wurde ein Score von ≥ 2,000, für 3 % (3/117) ein Score von ≥ 1,700 und für 2 % (2/117) ein Score von < 1,700 erzielt. In Bezug auf die nötigen Wiederholungen gelangen korrekte Identifizierungen mit einem Score von ≥ 2,000 im ersten Versuch für 80 % (93/117) mit der erweiterten Datenbank anstatt 54 % (63/117) der Stämme bzw. Isolate unter Nutzung der ursprünglichen Datenbank. Bei Nutzung zweier verschiedener Anzuchtmedien beeinflussten die Inhaltsstoffe der Medien an sich die Proteinspektren nicht direkt, jedoch waren wahrscheinlich auf Grund des verbesserten Wachstums, die erzielten Scorewerte mit AL-Avian Mycoplasma-Medien höher als bei SP4-Flüssigmedien. Die beispielhaft untersuchte Sensitivität der MALDI-TOF MS-Analyse ergab eine korrekte Speziesidentifizierung mit einem Score von ≥ 2,000 bei einer Bakterienmenge von 2,9 x 107 kbE Einheiten / ml für das untersuchte M. gallisepticum-Isolat und für 5,7 x 106 kbE Einheiten / ml für das M. gypis-Isolat, während ab 4,1 x 106 bzw. 5,6 x 105 kbE Einheiten / ml keine Identifizierung mehr möglich war.
Im Rahmen dieser Arbeit konnte gezeigt werden, dass MALDI-TOF MS in der Lage ist, aviäre Mykoplasmen zu identifizieren und in Bezug auf ihre Spezies zu differenzieren. MALDI-TOF MS stellt somit in Kombination mit einer entsprechenden Datenbank eine vielversprechende Methode zur schnellen und verlässlichen Identifizierung aviärer Mykoplasmenspezies dar.
In der vorliegenden Arbeit wurde die Anwendbarkeit von MALDI-TOF MS zur Identifizierung und Differenzierung aviärer Mykoplasmen evaluiert. Dazu wurde eigens eine massenspektrometrische Referenzdatenbank erstellt, welche anfänglich 27 MSP aus 23 aviären Mykoplasmenspezies beinhaltete. Nach Erstellung der Datenbank wurden 112 Feldisolate, sowie vier Lebendimpfstoff-Isolate und ein Referenzstamm aviärer Mykoplasmen, deren Spezies bereits mittels genusspezifischer PCR und Sequenzierung des Amplifikats bzw. einer speziesspezifischen PCR und Restriktionsenzymanalyse identifiziert worden waren, mit MALDI-TOF MS und der Mykoplasmen-Referenzdatenbank mit maximal 2 Wiederholungen untersucht.
Die MALDI-TOF MS-Analyse wies für 70 % (82/117) der Stämme bzw. Isolate einen Score von ≥ 2,000, für 20 % (23/117) einen Score von ≥1,700 und für 10 % (12/117) Identifizierungsergebnisse mit einem Score von < 1,700 auf. Es zeigte sich, dass besonders die Analysen der Isolate von M. gallisepticum, M. glycophilum, M. gypis, M. synoviae sowie M. sturni zu niedrigen Scores führten. Daraufhin wurde die MSP-Referenzdatenbank auf 36 MSP erweitert und hier besonders die genannten Spezies berücksichtigt. Der erneute Vergleich der bereits gemessenen Spektra mit der erweiterten Datenbank führte zu einer deutlichen Verbesserung der Ergebnisse. Für 96% (112/117) der Isolate bzw. Stämme wurde ein Score von ≥ 2,000, für 3 % (3/117) ein Score von ≥ 1,700 und für 2 % (2/117) ein Score von < 1,700 erzielt. In Bezug auf die nötigen Wiederholungen gelangen korrekte Identifizierungen mit einem Score von ≥ 2,000 im ersten Versuch für 80 % (93/117) mit der erweiterten Datenbank anstatt 54 % (63/117) der Stämme bzw. Isolate unter Nutzung der ursprünglichen Datenbank. Bei Nutzung zweier verschiedener Anzuchtmedien beeinflussten die Inhaltsstoffe der Medien an sich die Proteinspektren nicht direkt, jedoch waren wahrscheinlich auf Grund des verbesserten Wachstums, die erzielten Scorewerte mit AL-Avian Mycoplasma-Medien höher als bei SP4-Flüssigmedien. Die beispielhaft untersuchte Sensitivität der MALDI-TOF MS-Analyse ergab eine korrekte Speziesidentifizierung mit einem Score von ≥ 2,000 bei einer Bakterienmenge von 2,9 x 107 kbE Einheiten / ml für das untersuchte M. gallisepticum-Isolat und für 5,7 x 106 kbE Einheiten / ml für das M. gypis-Isolat, während ab 4,1 x 106 bzw. 5,6 x 105 kbE Einheiten / ml keine Identifizierung mehr möglich war.
Im Rahmen dieser Arbeit konnte gezeigt werden, dass MALDI-TOF MS in der Lage ist, aviäre Mykoplasmen zu identifizieren und in Bezug auf ihre Spezies zu differenzieren. MALDI-TOF MS stellt somit in Kombination mit einer entsprechenden Datenbank eine vielversprechende Methode zur schnellen und verlässlichen Identifizierung aviärer Mykoplasmenspezies dar.
| Erscheinungsdatum | 15.12.2020 |
|---|---|
| Reihe/Serie | Edition Scientifique |
| Sprache | deutsch |
| Maße | 146 x 210 mm |
| Gewicht | 300 g |
| Themenwelt | Veterinärmedizin |
| Schlagworte | Doktorarbeit • Uni • Wissenschaft |
| ISBN-10 | 3-8359-6914-5 / 3835969145 |
| ISBN-13 | 978-3-8359-6914-8 / 9783835969148 |
| Zustand | Neuware |
| Informationen gemäß Produktsicherheitsverordnung (GPSR) | |
| Haben Sie eine Frage zum Produkt? |
Mehr entdecken
aus dem Bereich
aus dem Bereich
Ein Handbuch für Tiermedizinische Fachangestellte
Buch | Softcover (2025)
Lehmanns Media (Verlag)
16,95 €
Buch | Hardcover (2025)
Schlütersche (Verlag)
89,00 €
Buch | Hardcover (2025)
John Wiley & Sons Inc (Verlag)
228,66 €